La Displasia dell'Anca...parte 4
Genetica, Ereditabilità e nuove frontiere
Quando si parla di genetica relativa alla Displasia dell’Anca (HD) sembra che ci si addentri in un ambito in cui non ci sono certezze ma solo oscuri misteri, in cui il ruolo dei geni è totalmente sconosciuto. Secondo alcuni, anche addetti ai lavori, gli stessi genetisti non saprebbero a che santo votarsi quando si parla di geni e HD, nel senso che non ci sarebbero informazioni certe sui geni coinvolti nella patologia e la loro trasmissione. E’ veramente così?
Non sarà inutile ricordare che, geneticamente parlando, l’HD è un tratto Quantitativo o Poligenico (Robinson, 1991): la sua espressione è dovuta ad una complessa interazione di molti geni, espressione genica a sua volta influenzata da fattori ambientali. In questo articolo prenderemo in esame solo la componente genetica, quella responsabile della trasmissione della patologia.
Nello studio dei Tratti Quantitativi applicati alla Selezione (in questo caso volta a ridurre la presenza del tratto) è di cruciale importanza considerare l’Ereditabilità, quel parametro che misura la facilità con cui i genitori trasmettono ai figli un determinato fenotipo. L’Ereditabilità è osservata e stimata in modo empirico da tempo immemore dagli allevatori; più recentemente, in ambito zootecnico, si compie la stima attraverso misurazioni fenotipiche e complessi calcoli statistico-matematici che forniscono un indice di Ereditabilità (h2) che si esprime in un valore compreso tra 0 e 1 e che può essere espresso anche in percentuale. (Pagnacco, 2004). Si usa considerare tre categorie di Ereditabilità, corrispondenti a dei range di valore applicati a h2. Si considerano come “Valore Elevato” i range compresi tra 0,5 e 0,8 (50%-80%), valori che si riscontrano in quei Tratti Quantitativi in cui il Fenotipo rispecchia bene il Genotipo e in cui l’ambiente ha un effetto limitato. Valori tra 0,2 e 0,5 (20%-50%) sono considerati “Valori Intermedi”, tipici di Tratti Quantitativi dove gli effetti genici sono mascherati in maniera considerevole dalle condizioni ambientali. In questi casi la scelta del Fenotipo non è sufficiente in ambito di Selezione per avere una risposta agli obiettivi prefissati. In ambito zootecnico si procede attraverso la valutazione della progenie o attraverso la stima di Indici Genetici (VGS) per valutare la capacità di trasmettere di un determinato Riproduttore. I “Valori Bassi” di Ereditabilità sono quelli compresi tra 0 e 0,2 (0-20%) che indicano un preponderante mascheramento dell’espressione genica da parte delle condizioni ambientali. La Selezione procede come per i tratti di “Valore Intermedio” ma la risposta è molto più lenta e in alcuni casi incerta. (Pagnacco, 2004).
Esistono studi di Ereditabilità sull’HD? Certamente. Prima di valutarne le stime, però, è opportuno fare una piccola premessa: la condizione fenotipica del cane displasico può essere data da molti parametri. Quattro sono le misure radiografiche che principalmente definiscono l’HD in base alle stime (Ostrander & Ruvinsky 2012), sono:
-DI (Distraction Index, tecnica che consente di valutare la lassità articolare)
-EHR (Ventrodorsal, Exended Hip Joint Radiograph, tecnica standard, OFA)
-DLS (Dorsolateral Subluxation Scores, tecnica che permette la valutazione della lassità passiva coxo- femorale)
-NA (Norberg Angulation, misura che permette di stimare la copertura dell’acetabolo sulla testa del femore)
L’Ereditabilità dell’HD è definita in senso generale come Valore Intermedio compreso tra 0,2 e 0,6 (Ostrander & Ruvinsky, 2012), nello specifico si attesta su valori di 0,2/0,3 per EHR (Breur et al., 2001; Hou et al., 2010) ma raggiunge valori di 0,5/0,6 su DI e DLS per popolazioni strettamente imparentate (Leighton, 1997; Todhunte et al., 2003).
Dato sicuramente interessante è la correlazione tra le misure: DI e DLS riflettono principalmente la lassità articolare mentre EHR e NA riflettono la conformazione ossea dell’articolazione. Le singole misure di ognuna di queste due coppie sono strettamente correlate tra loro geneticamente (0,9) ma solo moderatamente se si confrontano a coppie di valori (Zhang et al., 2009).
Parallelamente allo studio delle Ereditabilità, i tratti Quantitativi possono essere studiati e valutati in campo genomico attraverso l’individuazione dei QTLs (Quantitative Traits Loci) che altro non sono che tratti del DNA che contengono - o sono collegati - (Linkage) a geni che sono alla base di un Tratto Quantitativo.
Nell’ambito dell’HD sono stati individuati, attraverso numerosi studi, molti QTLs in diversi dei 38 cromosomi (CFA):
-CFA03 (Chase et al., 2005)
-CFA04, 09, 10, 11, 16, 22, 25, 29, 30, 35 (Todhunter et al., 2005)
-CFA01, 03,04, 08, 09, 16, 19, 26, 33 (Marschall & Distl, 2007)
Chase et al. (2004) ha identificato due distinti QTLs in CFA01 associati a HD correlata a NA (angolo di Norberg), nel Cao de Agua Portoghese. Lo stesso cromosoma (CFA01) ospita QTLs per HD con misurazione di NA nel Labrador Retriever, insieme ad altri QTLs in CFA02, 10, 20, 22, 32 (Phavaphutanon et al., 2009). QTLs per HD è stato identificato in CFA01 anche per il Pastore Tedesco (Marschall & Distl, 2007), QTLs per HD in generale per Labrador Retriever e Pastore Tedesco in CFA04, 09, 10, 16, 22.
L’identificazione di QTLs per HD negli stessi cromosomi in studi indipendenti su diverse razze dimostra il fatto che siano identici per discendenza ereditaria (Ostrander & Ruvinsky, 2012).
Un importante studio Genomico (SNPs) su HD e OA (Osteoartrite) che ha visto coinvolti un totale di 721 cani di razza - Labrador Retriever, Greyhound, Pastore Tedesco, Golden Retriever, Rottweiler, Border Collie, Alano - ha indentificato 4 Quantitative Trait Nucleotides (QTNs, polimorfismi di singoli nucleotidi per Tratti Quantitativi) associati a misure dell’angolo di Norberg in cani con Displasia. Lo stesso studio, interessante particolare da rilevare, ha associato basi genetiche omologhe per disturbi del cane e dell’uomo (Zhou Z., 2010).
Ad un altro studio genomico si deve l’identificazione di una mutazione, grazie a fine sequenziamento genomico (SNPs) di 100 Labrador Retriever, del gene FBN2 (che regola la fibrillina, proteina che costituisce le microfibrille che hanno funzione regolatrice e strutturale) nel cromosoma CFA11 come strettamente correlata al peggioramento della patologia (Friedenber S.G. et al., 2008). Un interessante e recente studio comparativo per 9 patologie (HD e DM quelle più note) in diverse razze della popolazione canina di Belgio, Olanda e Germania ha identificato questa mutazione del gene FNB2 in 49 di esse. In 44 razze, una di queste patologie è stata identificata per la prima volta. Per 10 razze, tra cui il Saarloos, la mutazione è stata identificata come allele unico (Broeckx et al., 2013).
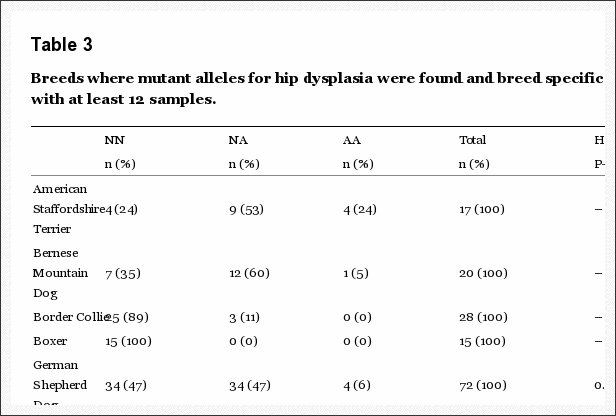
Il dato più innovativo viene da uno studio, pubblicato nel 2011 con il titolo: “Canine Hip Dysplasia is predictable by Genotyping” (“La Displasia dell’Anca del cane è prevedibile attraverso la genotipizzazione”): sono stati presi due set di cani appartenenti a 6 razze e genotipizzati con pannelli SNPs ad alta intensità che coprono l’intero genoma. Il primo set comprendeva 359 cani, su cui fu calcolata una formula di valore genomico (GBV Genomic Breeding Value) in base a EBV (Estimated Breeding Value, i famigerati VGS o Valori Genetici Stimati) dell’angolo di Norberg e i loro genotipi. Per studiare come la formula funzioni su ogni singolo cane è stata eseguita una validazione incrociata mascherando EBV in ogni test, per ogni cane. I dati genomici e EBV dei restanti cani sono stati usati per stimare il GBV del cane con EBV mascherato. In questo secondo set di cani furono inclusi 38 Labrador Retriever senza rapporti di parentela con i cani della stessa razza presenti nel primo set. L’analisi incrociata ha evidenziato una forte correlazione (R > 0,7) tra EBV e GBV, dimostrando ancora una volta la validità dei Valori Genetici Stimati nel calcolo dell’ereditabilità di Tratti Quantitativi come HD. Una moderata correlazione (R > 0,5) è stata evidenziata tra l’Angolo di Norberg osservato e l’angolo di Norberg stimato attraverso GBV. La sensibilità specifica di ogni valore predittivo è stata sempre sopra il 70%.
Riporto per intero le conclusioni tratte alla fine dell’articolo scientifico:
“Prevedere la HD dai dati genomici è fattibile, e ciò può essere applicato per la gestione del rischio di HD e di selezione precoce per il miglioramento genetico per ridurre la prevalenza di malattia HD nei programmi di allevamento. La previsione può essere attuata prima della scadenza classica, alla quale età sono tradizionalmente applicati gli attuali programmi di screening radiografici, appena il DNA è disponibile.” (G. Guo et al., 2011)
Appare chiaro che le informazioni genetiche sulla componente ereditaria della Displasia dell’Anca sono ben lungi dall’essere un mistero, anzi: sono in continuo incremento, la loro applicazione in ambito di specifici studi di razza è una possibilità concreta che è in uso da molto tempo in quegli ambiti cinofili che sono più evoluti e sensibili. Se appaiono così distanti è perché le Società Specializzate, che dovrebbero occuparsi (da statuto) del miglioramento genetico delle razze, sono prive della volontà di perseguire obiettivi importanti in questo senso, limitandosi all’organizzazione degli eventi espositivi e poco altro. Considerando il non trascurabile livello di screening della nostra razza (solo il Database di CLC.ITALIA.it presenta oltre 1300 risultati ufficiali di HD) sarebbe possibile pianificare le basi per importanti iniziative sullo studio e la prevenzione della Displasia dell’Anca, tenendo conto del fatto che esistono in Italia tecnologie SNPs (sequenziatori Illumina) utilizzabili in ambito zootecnico e cinofilo, nello specifico. I costi non sono certo esigui ma in continua discesa. Nel 2012, il costo di una sequenza su pannello Canine Illumina HD da 174.000 SNPs, mi veniva offerto - come rappresentante di CLUB - a 200 euro ad analisi (per ogni singolo cane) quando solo due anni prima l’utilizzo delle stesse tecnologie al di fuori della Genetica Forense, Zootecnia da Reddito e Ricerca Scientifica era impensabile per i costi ingenti. Considerando che, sulla carta, l’ENCI ha un fondo di oltre 100.000 euro accantonati per ricerche e progetti delle Società Specializzate è impossibile non rammaricarsi per l’assoluto immobilismo nella nostra razza.
Alessio Camatta
Bibliografia:
-Roy Robinson “Genetics for the Dog Breeder” 1991
-Giulio Pagnacco “Genetica Animale Applicata” 2004
-Aldo Vezzoni “Early Diagnosis of canine Hip Dysplasia” 2005
- A. Brunetti, A. Greco “L’esame TC nella Displasia dell’Anca del Cane” 2002-2005
-Todhunter R.J. "Quantitative Trait Loci for Hip Dysplasia in a cross-breed canine pedigree" 2005
-Chase K. "Genetic regulation of Osteoarthtritis: a Qtl regulating cranial and caudal acetabular osteophite formation
in the hip joint of the dog" (2005)
-Marschall, Y. Distl, O. "Mapping quantitative trait loci for canine Hip Dysplasia in German Sheperd Dogs" 2007
-Friedenber S.G. “Evaluation of a fibrilin 2 haplotype associated with canine hip dysplasia and..ostheoarthrtis in dog.” 2008
- Zhengkui Zhou “Differential Genetic Regulation of Canine Hip Dysplasia and Osteoarthritis” 2010
-G. Guo “Canine hip dysplasia is predictable by genotyping” 2011
-Ostrander, Ruvinsky “The Genetics of the Dog” 2012
- Bart J.B. Broeckx “The Prevalence of Nine Genetic Disorders in a Dog Population from Belgium, the Netherlands...” 2013
Articolo inserito il 29/01/2014





